Перейти к:
Циркулирующие микроРНК — перспективные биомаркеры для оценки риска развития антипсихотик-индуцированного метаболического синдрома (обзор): часть 2
https://doi.org/10.30895/2312-7821-2025-499
Резюме
ВВЕДЕНИЕ. В первой части статьи был рассмотрен антипсихотик-индуцированный метаболический синдром (АИМетС) как распространенная нежелательная реакция при фармакотерапии психических расстройств и болезней зависимости. Показаны подходы к спектру и оценке основных и дополнительных клинических и лабораторных маркеров метаболического синдрома (МетС) у пациентов с расстройствами шизофренического спектра в целом и АИМетС в частности. Изменение уровня экспрессии циркулирующих микроРНК в крови может рассматриваться как одна из перспективных методологий прогнозирования и диагностики АИМетС.
ЦЕЛЬ. Рассмотреть роль циркулирующих микроРНК как эпигенетических биомаркеров развития основных звеньев патогенеза АИМетС.
ОБСУЖДЕНИЕ. Проведен анализ и систематизация результатов фундаментальных и клинических исследований роли циркулирующих микроРНК, влияющих на основные звенья патогенеза и прогрессирования АИМетС, опубликованных в период 2012–2024 гг. Проанализированы результаты исследований, отражающих роль микроРНК в ключевых звеньях патогенеза МетС и АИМетС: окислительном стрессе, системном воспалении, регуляции адипогенеза и развитии центрального ожирения, липидного метаболизма, гомеостаза холестерина липопротеинов высокой/низкой плотности, атерогенеза, жировом гепатозе, а также регуляции чувствительности к инсулину, его экспрессии, метаболизма глюкозы, аппетита, экспрессии нейропептида Y, орексина тиреоидных гормонов, паратиреоидного гормона, чувствительности к лептину. Показано, что персонализированная оценка безопасности фармакотерапии может зависеть от паттерна циркулирующих микроРНК, которые индуцируют или ингибируют основные звенья патогенеза АИМетС. Различия в результатах проанализированных исследований микроРНК могут быть обусловлены тем, что фундаментальные (преимущественно) и клинические исследования имели вариабельный дизайн, а также тем, что в них не учитывались другие модифицируемые и немодифицируемые факторы риска АИМетС. Предложена градация микроРНК по степени риска развития АИМетС.
ВЫВОДЫ. Этот обзор демонстрирует, что чувствительность и специфичность эпигенетических биомаркеров АИМетС могут варьировать в широком диапазоне в зависимости от характера их влияния (предиктивного или протективного) на один или несколько основных звеньев патогенеза рассматриваемой распространенной нежелательной реакции психофармакотерапии. Наиболее изученными являются микроРНК — предиктивные биомаркеры окислительного стресса (miR-1, miR-21, miR-23b, miR-27a) и системного воспаления (miR-21, miR-23a, miR-27a) у пациентов с высоким риском развития МетС и АИМетС. Перспективными эпигенетическими биомаркерами АИМетС являются микроРНК, влияющие на уровень экспрессии нейропептидов и чувствительность к ним, включая нейропептид Y (miR-let7b, miR-29b, miR-33 и др.), лептин (miR-let7a, miR-9, miR-30e и др.) и орексин (miR-137, miR-637, miR-654 и др.).
Ключевые слова
Для цитирования:
Шнайдер Н.А., Насырова Р.Ф., Пекарец Н.А., Гречкина В.В., Петрова М.М. Циркулирующие микроРНК — перспективные биомаркеры для оценки риска развития антипсихотик-индуцированного метаболического синдрома (обзор): часть 2. Безопасность и риск фармакотерапии. 2025;13(4):394-410. https://doi.org/10.30895/2312-7821-2025-499
For citation:
Shnayder N.A., Nasyrova R.F., Pekarets N.A., Grechkina V.V., Petrova M.M. Circulating MicroRNAs Are Promising Biomarkers for Assessing the Risk of Antipsychotic-Induced Metabolic Syndrome (Review): Part 2. Safety and Risk of Pharmacotherapy. 2025;13(4):394-410. https://doi.org/10.30895/2312-7821-2025-499
ВВЕДЕНИЕ
Расстройства шизофренического спектра (РШС) относятся к распространенным социально значимым психическим расстройствам, связанным с сокращением продолжительности жизни [1][2]. В последние годы продемонстрирована роль антипсихотик-индуцированного метаболического синдрома (АИМетС) у пациентов с РШС как одной из основных причин смерти этих пациентов [3]. Повышение безопасности фармакотерапии антипсихотиками (АП) и оценка риска развития АИМетС и ассоциированных сердечно-сосудистых заболеваний являются одной из приоритетных стратегий ведения пациентов с РШС [4], включая поиск и внедрение в реальную клиническую практику новых лабораторных биомаркеров АИМетС [5].
Малые некодирующие рибонуклеиновые кислоты (микроРНК) играют большую роль в регуляции различных физиологических и патологических процессов, участвующих в механизмах развития АИМетС у пациентов с РШС. К таким механизмам относятся основные звенья патогенеза АИМетС: окислительный (оксидативный) стресс [6][7], системное воспаление [8][9], дифференцировка адипоцитов и центральное ожирение [8][10][11], метаболизм липидов и глюкозы [8][11–23], регуляция аппетита [24–28], уровень нейропептида Y (NPY) [24][29][30], чувствительность к лептину [9][28][30], уровни орексина [31][32], тестостерона [33], тиреоидных [34] и паратиреоидного [35] гормонов.
В первой части настоящего обзора [36] было показано, что детекция изменений уровня экспрессии циркулирующих микроРНК в доступных образцах (кровь: сыворотка, экзосомы, мононуклеары) как эпигенетических биомаркеров АИМетС перспективна как одна из альтернативных методологий прогнозирования и диагностики рассматриваемой нежелательной реакции у пациентов с РШС по сравнению с генетическими биомаркерами (образцами ДНК). Циркулирующие микроРНК характеризуются стабильностью при хранении образцов (в том числе при многократных циклах замораживания и оттаивания), воспроизводимостью и согласованностью сигнатур у отдельных пациентов [36].
Для понимания механизмов метаболических изменений необходимо более подробно рассмотреть предиктивные микроРНК, ассоциированные с повышением риска основных звеньев патогенеза АИМетС у пациентов с РШС.
Цель работы — рассмотреть роль циркулирующих микроРНК как эпигенетических биомаркеров развития основных звеньев патогенеза антипсихотик-индуцированного метаболического синдрома.
Методика поиска литературы описана в первой части обзора [36].
ОСНОВНАЯ ЧАСТЬ
МикроРНК как эпигенетические биомаркеры основных звеньев патогенеза АИМетС
Окислительный стресс
Имеются многочисленные клинические и экспериментальные отчеты, демонстрирующие изменения, индуцированные приемом АП, со стороны оксидантно-антиоксидантного баланса у пациентов с РШС [37–40]. АП первого (галоперидол) и второго (клозапин, оланзапин, рисперидон) поколения индуцируют значительное снижение активности супероксиддисмутазы и каталазы, активируют перекисное окисление липидов и образование активных форм кислорода (АФК) [38]. Сигнальный путь эндогенного ядерного фактора 2, связанного с эритроидным фактором 2 (Nrf2), участвует в механизме защиты от окислительного стресса и играет важную роль как в развитии РШС, так и в развитии АИМетС, но сложный баланс Nrf2 с NF-κВ (ядерным фактором, усиливающим легкую цепь каппа активированных В-клеток) и его перекрестные помехи с фактором транскрипции Nrf2 имеют решающее значение при тяжелом окислительном стрессе у пациентов с РШС [39].
К микроРНК, индуцирующим окислительный стресс, относят miR-1, miR-21, mir-23b, miR-27a, miR-28, miR-29, miR-34a, miR-92a, miR-93, miR-101, miR-106b, miR-128, miR-129-3p, miR-140, miR-142, miR-144, miR-146, miR-148, miR-153, miR-155, miR-181c, miR-193b, miR-320, miR-365, miR-375, miR-383, miR-495, miR-503, miR-802. Механизмы индукции окислительного стресса имеют связь с сигнальным путем Nrf2 и обладают высокой степенью контроля над этим путем на разных стадиях его развития. NFE2L2/Nrf2 является критическим биомаркером, ассоциированным с цитопротекторными реакциями в ответ на окислительные и электрофильные нарушения. Nrf2 убиквитинируется и направляется на деградацию келч-подобным ECH-ассоциированным белком 1 (Keap1), в то время как путь Keap1/Nrf2 представляет собой наиболее важный цитопротекторный путь, реагирующий на повышение уровня АФК [41]. МикроРНК участвуют в контроле сигнального пути Nrf2 посредством нескольких механизмов: изменение ядерной транслокации Nrf2; влияние на экспрессию Nrf2; контроль медиаторов, расположенных выше по потоку Nrf2; модулирование Keap1.
Сиртуин 1 (Sirt1) — это НАД+-зависимая деацетилаза, которая является важным антиоксидантным ферментом, связанным с развитием сахарного диабета. Sirt1 использует первый путь, PGC1a/ERRa, чтобы индуцировать Nrf2, хотя он также способен напрямую активировать Nrf2, используя другие механизмы. Например, у пациентов с оланзапин-индуцированным МетС наблюдались заметно более низкие уровни Sirt1 в плазме крови, чем у пациентов c шизофренией без МетС и здорового контроля [40]. Гораздо менее изученным является сиртуин 3 (Sirt3) [42], который положительно регулирует экспрессию фактора транскрипции O1 и О3 (forkhead box protein, FOXO1/3), что приводит к антиоксидантному ответу [7]. Sirt3 экспрессируется в тканях и органах с высокой скоростью метаболизма, включая печень, головной мозг, сердце и бурую жировую ткань, и играет важную роль в регуляции окислительного стресса и митохондриального метаболизма [43], поэтому изучение влияния микроРНК на уровень экспрессии этой молекулы важно с научной и клинической точек зрения.
Так, miR-23b индуцирует окислительный стресс через снижение экспрессии Sirt1 и Nrf2 [7][44]. При гиперэкспрессии miR-34a происходит подавление экспрессии гена, кодирующего Nrf2, с заметным снижением уровней мРНК и белка Nrf2. В то же время при подавлении экспрессии miR-34a восстанавливается Nrf2-зависимый антиоксидантный путь [6][45]. MiR-92a ингибирует Sirt1 и эндотелиальную синтазу оксида азота (eNOS) [46], а также активирует инфламмасомы, что усугубляет дисфункцию эндотелия при окислительном стрессе [7]. MiR-128 снижает уровень экспрессии Sirt1 и уровни экспрессии белка Nrf2, способствуя развитию окислительного стресса [6][7]. MiR-140 непосредственно воздействует на уровни Nrf2 и Sirt2, изменяя уровни экспрессии HO-1, NQO1, Gst, GCLM, Keap1 и FOXO3a [6].
MiR-27а, miR-142, miR-144 и miR-153 являются регуляторными микроРНК для Nrf2, снижая ее транскрипцию [6]. Активация и экспрессия сигнального пути Nrf2 и его нижестоящего медиатора HO-1 заметно ускоряются при подавлении экспрессии miR-320 [6]. MiR-383 опосредует окислительный стресс и апоптоз, подавляя пероксиредоксин 3 типа (PRDX3) [7].
Системное воспаление
Известно, что типичные и атипичные АП влияют на выработку провоспалительных цитокинов [47]. Например, галоперидол может изменять уровень интерферона гамма (ИФ-γ), фактора некроза опухоли альфа (ФНО-α), выработку интерлейкинов (ИЛ-2, ИЛ-10, ИЛ-237) и др., а клозапин, рисперидон и кветиапин (помимо вышеописанных цитокинов) также изменяют выработку ИЛ-4, ИЛ-104, ИЛ-646 [48]. Исследования, посвященные оценке взаимосвязей между приемом АП и изменением биомаркеров системного воспалительного ответа, убедительно демонстрируют изменение уровней этих биомаркеров в периферической крови, но их уровень и влияние на течение психических расстройств и риск развития АИМетС все еще являются предметом дискуссий [49][50].
Системное воспаление характеризуется повышением уровней сывороточных белков острой фазы и провоспалительных цитокинов, включая С-реактивный белок, ФНО-α, ИЛ-1β, ИЛ-6 и ИЛ-17, а также инфильтрацией макрофагов и Т-лимфоцитов в инсулинозависимых тканях [8]. В целом микроРНК регулируют различные стадии воспаления, начиная с инициации, альтерации, экссудации, пролиферации и разрешения, посредством как положительной, так и отрицательной обратной связи. При положительной обратной связи набор событий ограничивает не только инвазию патогенов, но и успешное восстановление поврежденных тканей. Напротив, отрицательная обратная связь, активируемая при тяжелом воспалении, помогает поддерживать тканевой гомеостаз.
В последние годы идентифицировали микроРНК (miR-21, miR-23a, miR-27a, miR-29a, miR-34a, miR-34c, miR-92a, miR-132, miR-138, miR-155, miR-200, miR-let7a), способные реализовывать системное воспаление через их измененную экспрессию в определенных иммунных клетках. Как часть воспалительной реакции экспрессия микроРНК часто регулируется на разных стадиях, таких как синтез, процессинг и стабилизация пре- или зрелых микроРНК [8][9].
Установлено, что повышенный уровень miR-let7a активирует провоспалительный путь NF-κB посредством нацеливания на ингибитор ядерного фактора-κB (IκB) киназы. Повышенный уровень miR-21 в жировой ткани характерен для людей с ожирением и сахарным диабетом 2 типа, являющимся, по сути, ведущим клиническим проявлением МетС, ассоциированным c вялотекущим системным воспалением, усилением адипогенной дифференцировки посредством модуляции передачи сигналов трансформирующего фактора роста бета-1 (TGF-β1). Также miR-21 играет решающую роль в ангиогенезе посредством регуляции VEGFA [9][51][52].
Провоспалительный механизм miR-23a связан с активацией в M1-макрофагах провоспалительного пути NF-κB с одновременным ингибированием противовоспалительного пути JAK1/STAT6 [9][53]. Дефицит антиоксидантного Sirt1 индуцирует гиперэкспрессию miR-132 в первичных преадипоцитах человека, что индуцирует высвобождение провоспалительных цитокинов. Семейство miR-34 (miR-34a и miR-34c) индуцирует высвобождение провоспалительных цитокинов и хемокинов посредством нацеливания на G-белок (LGR4), тем самым задерживается локальный воспалительный ответ [9][54].
Гиперэкспрессия провоспалительной miR-92a приводит к усилению регуляции нескольких генов, кодирующих провоспалительные цитокины в макрофагах-реципиентах [9], а гиперэкспрессия miR-138 и miR-155 способствует системному воспалению путем активации провоспалительного сигнального пути NF-κB, а также путей MyD88 и TRIF, способствующих повышению уровней провоспалительных цитокинов и супрессии цитопротективного белка Sirt1 [9][55].
МiR-200 демонстрирует провоспалительный ответ путем ее влияния на синтез белка Zeb-1, участвующего в повышении активности фермента циклооксигеназы-2 (COX-2) и моноцитарного хемотаксического протеина-1 (MCP-1) в гладкомышечных клетках сосудов при сахарном диабете 2 типа [9]. Этот путь также является важным при развитии МетС у пациентов с психическими расстройствами, получающих АП [56].
Регуляция адипогенеза и развитие центрального ожирения
Адипогенез является процессом, посредством которого адипоциты развиваются из стволовых клеток жирового происхождения с образованием жировой ткани [57]. Нарушение адипогенеза и регуляции метаболизма жировой ткани ассоциированы с различными проявлениями АИМетС (ожирение, сахарный диабет 2 типа, сердечно-сосудистые заболевания). Понимание механизма формирования, функций, состава секретома адипоцитов важно для разработки таргетной терапии МетС и АИМетС у пациентов с РШС [58]. АП первой линии (галоперидол и др.) и новых поколений (оланзапин, клозапин, рисперидон и др.) могут индуцировать адипогенез, что сопровождается гипоэкспрессией белков, связывающих стероид-регулирующие элементы 1 (SREBP1), SREBP2, синтазы жирных кислот (FAS), рецепторы липопротеинов низкой плотности (ЛПНП) и гидроксиметилглутарил-кофермент А-редуктазы (HMGR), гена PPARG, кодирующего рецептор-γ активированного пролифератора пероксисом [59]. Кроме того, клозапин и оланзапин усиливают дифференцировку преадипоцитов 3T3-L1 в зрелые адипоциты за счет активации пути SREBP1 [60].
МикроРНК способны оказывать непосредственное регулирующее действие на адипогенез. В ходе исследований установлено индуцирующее влияние на адипогенез и развитие центрального ожирения для ряда микроРНК (miR-17, miR-20a, miR-21, miR-103, miR-128-1, miR-143, miR-144, miR-146b, miR-148a, miR-194, miR-210, miR-322, miR-375, intronic miR-378). Одним из механизмов микроРНК опосредованной регуляции адипогенеза является регуляция экспрессии трансформирующего фактора роста-β (TGF-β) — белка, регулирующего пролиферацию, дифференцировку и рост адипоцитов и модулирующего экспрессию и активацию других факторов роста, включая ИФ-γ и ФНО-α [58].
Так, miR-21 стимулирует адипогенез, модулируя передачу сигналов преадипоцитов TGF-β1. Усиление адипогенной дифференцировки в преадипоцитах 3T3-L1 связано с повышенной экспрессией miR-17, miR-21a, miR-21 и miR-143, стимулирующих экспрессию адипогенного фактора транскрипции C/EBPα и усиливающих передачу сигналов TGF-β [8][10].
MiR-103 удваивает экспрессию адипогенного фактора транскрипции PPARγ, также увеличивая экспрессию белка, связывающего жирные кислоты 4 (FABP4), и адипонектина примерно в 9 и 4 раза соответственно [10]. Также miR-128-1 регулирует гомеостаз циркулирующих липопротеинов, а также экспрессию генов, кодирующих PPAR, и других регуляторов окисления жирных кислот и системного воспаления, приводя к развитию центрального ожирения [11].
MiR-144 снижает экспресcию фактора транскрипции FOXO1, подавляя его стимулирующее действие на адипонектин, тем самым ослабляя ингибирующее действие адипонектина на адипогенез в преадипоцитах 3T3-L1, а miR-146b способствует адипогенезу в преадипоцитах 3T3-L1 посредством подавления экспрессии Sirt1 [10].
Группа микроРНК, таких как miR-148a, miR-194, miR-210 и miR-322, индуцирует адипогенез путем подавления передачи сигналов пути Wnt/β-катенина — ключевого ингибитора адипогенеза [10][61].
Гиперэкспрессия miR-375 инициирует дифференцировку адипоцитов путем увеличения уровней C/EBPα и PPARγ и одновременной индукции FABP4 адипоцитов и накопления триглицеридов. Гиперэкспрессия miR-375 способствует адипогенезу через дифференцировку преадипоцитов 3T3-L1 и подавление фосфорилирования внеклеточной протеинкиназы, регулируемой сигналом (ERK1/2) [10].
Регуляция липидного метаболизма
Различные механизмы, лежащие в основе нарушения регуляции метаболизма липидов, вызванных приемом АП различных поколений (включая АП-индуцированную гиперхолестеринемию и повышение риска развития атеросклероза у пациентов с РШС, в том числе взрослых и подростков) продолжают активно изучаться [62][63]. Так, A. Delacrétaz и соавт. [64] продемонстрировали, что у 49% подростков с РШС в течение первого месяца приема АП наблюдалось раннее повышение уровня общего холестерина на 5% и более, а у 33% в течение первого года лечения развилась гиперхолестеринемия. У пациентов со снижением уровня липопротеинов высокой плотности (ЛПВП) на ≥5% от исходного в течение первого месяца приема АП через 3 месяца лечения наблюдалось более значительное снижение этого биомаркера по сравнению с пациентами, у которых его уровень снизился менее чем на 5%.
Известно, что микроРНК оказывают непосредственное влияние на обмен липидов. Ингибируют липидный метаболизм miR-30c, miR-33a, miR-33b, miR-34a, miR-128-1, miR-144, miR-148a, miR-223, miR-246b.
MiR-34a опосредует реакцию печени на метаболический стресс, связанный с перегрузкой липидами, посредством подавления экспрессии ядерного фактора транскрипции гепатоцитов 4A (HNF4A), который является критическим фактором транскрипции в липидном обмене. Еще одним регуляторным центром, который подавляет гены, участвующие в биосинтезе холестерина, является мiR-223. Кроме того, miR-223 подавляет экспрессию гепатоцитами рецептора-поглотителя SR-BI, который транспортирует холестерин из ЛПВП в печень. Конечным результатом действия miR-223 является снижение уровня холестерина в печени [11].
MiR-246b воздействует на мРНК β-рецептора тиреоидного гормона (Trβ) в печени, что приводит к изменениям в экспрессии генов, отвечающих за липидный обмен, и снижению содержания внутриклеточных липидов [34].
Механизмы действия miR-30c, miR-33a, miR-33b, miR-128-1, miR-144 и miR-148a будут описаны далее, так как основной точкой приложения является регуляция гомеостаза холестерина ЛПНП и ЛПВП [11–15].
Регуляция гомеостаза холестерина ЛПВП
Прием АП первого и новых поколений нарушает гомеостаз ЛПВП и приводит к снижению их уровня в сыворотке крови [65]. В популяционном исследовании A. Richards-Belle и соавт. (2023) с участием 3255 пациентов показано, что применение АП было достоверно связано с более низким сывороточным уровнем ЛПВП и более высоким уровнем триглицеридов [66].
В последние годы установлено, что miR-33a и miR-33b координированно ингибируют обратный транспорт холестерина с периферии обратно в печень за счет подавления АТФ-связующих кассетных транспортеров A1 (ABCA1), а также ингибируют выведение холестерина из организма путем супрессии белка транспортера ABCB11 и липид-транспортирующей АТФазы 8B1 (ATP8B1), способствующих выведению холестерина из печени в желчь. Они также подавляют ферменты окисления жирных кислот, что приводит к снижению обмена липидов в клетках [11]. Эффекты miR-128-1, miR-144 и miR-148a ассоциированы с подавлением экспрессии ABCA1. Суммарный эффект этих микроРНК заключается в снижении оттока холестерина в ЛПВП [13][14], что делает их перспективными эпигенетическими биомаркерами АП-индуцированной дислипидемии.
Регуляция гомеостаза холестерина ЛПНП
Ассоциированные со снижением уровня ЛПВП микроРНК (miR-128-1, miR-148a) способствуют повышению уровня ЛПНП посредством подавления экспрессии их рецепторов, через которые периферические клетки поглощают липиды из циркулирующих ЛПНП. Так, ингибирование miR-128-1 и miR-148a у мышей приводило к усилению экспрессии рецепторов ЛПНП и увеличивало клиренс циркулирующих ЛПНП, что в последующем приводило к снижению их уровня в плазме [13].
Регуляция атерогенеза
Атерогенез является важным звеном патогенеза МетС и АИМетС. Показано, что длительный прием некоторых атипичных АП может повышать риск развития атеросклероза и ассоциированных с ним сердечно-сосудистых заболеваний у пациентов с РШС [67]. В последние годы продемонстрированно, что микроРНК, помимо ранее описанных механизмов влияния на липидный метаболизм и адипогенез, регулируют процессы атерогенеза. Так, ингибирование miR-33 увеличивает транспорт холестерина из макрофагов в плазму, печень и кал более чем на 80%, что может предотвращать образование пенистых клеток и атеросклероз за счет увеличения активности ABCA1 и, как следствие, повышения уровня ЛПВП. Гипоэкспрессия miR-33 взаимосвязана и с другими атеропротекторными эффектами, включая регуляцию функциональной поляризации макрофагов [11].
К сильным ингибиторам оттока холестерина из различных тканей, включая макрофаги, относится также miR-144. Так, показано, что гипоэкспрессия miR-144 на животных моделях приводит к замедлению прогрессирования атеросклероза [14].
Жировой гепатоз
Неалкогольная жировая болезнь печени (НАЖБП) является наиболее распространенной причиной хронических заболеваний печени в мире и одним из многих проявлений инсулинорезистентности и МетС [68]. Это прогрессирующее заболевание, которое характеризуется резистентностью печени к инсулину и воспалением, связанным с накоплением жира в печени. У пациентов, принимающих АП, независимо от клинического диагноза частота встречаемости НАЖБП выше, чем в общей популяции [68]. Гиперэкспрессия miR-34a (как у человека, так и у мышей) способствует развитию НАЖБП. Ингибирование этой микроРНК на мышиной модели оказывало терапевтический эффект при НАЖБП, что было связано с miR-34a-опосредованной репрессией PPARα и последующим подавлением метаболизма жирных кислот в печени [11].
Регуляция чувствительности к инсулину
Известно, что длительный прием АП может оказывать негативное влияние на рецепторы к инсулину и различные сигнальные инсулиновые пути. Как следствие, повышается риск развития инсулинорезистентности как одного из ведущих звеньев патогенеза АИМетС у пациентов с РШС [69–71]. Ряд микроРНК, таких как miR-let7, miR-15b, miR-19, miR-29, miR-33a/b, miR-103, miR-107, miR-143, miR-155, miR-223 miR-378, miR-451-1, miR-802, снижают чувствительность клеток различных органов и тканей к инсулину.
Высоко консервативная miR-let7 экспрессируется в скелетных мышцах, где она ингибирует рецептор инсулина 2 (IRS2) и рецептор инсулиноподобного фактора роста 1 (IGF1), тем самым снижая чувствительность мышечной ткани к инсулину [11].
Гиперэкспрессия miR-15b блокирует рецепторы инсулина IRS1 в гепатоцитах, способствуя развитию печеночной инсулинорезистентности [11]. МiR-19 ингибирует липидный гомолог фосфатазы и тензина (PTEN), участвующий в передаче сигналов инсулина в печени [11][ 72]. МикроРНК семейства miR-33 блокируют субстрат IRS2, который является ключевым компонентом инсулинового сигнального пути [11]. В адипоцитах miR-103 и miR-107 подавляют экспрессию кавеолина, что приводит к ослаблению силы сигналов, передаваемых на инсулиновые рецепторы [16].
Гиперэкспрессия miR-143 вызывает инсулинорезистентность за счет ингибирования ORP8 (OSBP-related protein 8), являющегося членом семейства оксистерол-строительного белка (OSBP), и блокады активации пути PI3K/AKT инсулином [11][ 73]. Секретируемая макрофагами циркулирующая miR-155 ингибирует передачу сигнала инсулина, подавляя экспрессию PPARγ в миоцитах (скелетных мышц и миокарда) и гепатоцитах [11][ 74].
Гены IRS1 (insulin receptor substrate 1 — субстрат 1 инсулинового рецептора) и SLC2A4 (solute сarrier family 2, member 4 — семейство переносчиков растворенных веществ 2 типа, член 4; известный также как GLUT4 — ген транспортера глюкозы 4 типа), участвующие в регуляции метаболизма глюкозы, являются мишенями для miR-223. Эта микроРНК также модулирует экспрессию регулируемого инсулином белка-переносчика GLUT4. Нарушение регуляции экспрессии этой микроРНК может подавлять сигнальный каскад инсулина, что может привести к инсулинорезистентности и сахарному диабету 2 типа [8].
MiR-378 подавляет экспрессию каталитической субъединицы фосфоинозитида 3-киназы (PI3K) p110a. Поскольку PI3K является ключевым трансдуктором инсулиновой сигнализации, устойчивая гиперэкспрессия miR-378 приводит к инсулинорезистентности [17], а miR-802 воздействует на экспрессию фактора транскрипции HNF1B, усиливая регуляцию HNF1B-ассоциированных генов SOCS1 и SOCS3 (кодирующих супрессоры 1 и 3 типа цитокинового сигнального пути), что ухудшает передачу сигнала инсулина путем ингибирования фосфорилирования белков IRS [18].
Регуляция экспрессии инсулина
АП могут блокировать мускариновые M3-рецепторы на мембране β-клеток островков Лангерганса поджелудочной железы, что приводит к подавлению секреции инсулина, стимулируемой холинергическими рецепторами, и препятствует транспортировке глюкозы в периферические ткани. Считается, что дефицит функции β-клеток, вызванный длительным приемом АП, обусловлен сочетанием инсулинорезистентности и сниженной секреции инсулина, стимулируемой глюкозой [75]. Экспрессия инсулина, а также слияние и высвобождение гранул инсулина, контролируется микроРНК.
МикроРНК, ингибирующими экспрессию и секрецию инсулина, являются: miR-7a, miR-26a, miR-29, miR-124a, miR-130a, miR-130b, miR-152, miR-187, miR-200, miR-204, miR-375 и miR-802.
Так, miR-7a ингибирует транскрипцию гена инсулина, подавляя экспрессию парного боксового белка Pax6 (paired box protein Pax-6, также известен как aniridia type II protein (AN2) — белок аниридии II типа или окулоромбин), а также ингибирует транскрипцию гена, кодирующего инсулиноподобный пептид Ilp2, посредством неустановленного механизма. В дополнение к регуляции экспрессии инсулина miR-7a снижает количество инсулина, выделяемого β-клетками островков Лангерганса поджелудочной железы, подавляя белки, участвующие в перестройке цитоскелета. Эта микроРНК подавляет экспрессию гена CAPZA1 (capping protein (actin filament) muscle Z-line, alpha 1), кодирующего альфа-1 субъединицу белка, покрывающего F-актин, необходимую для высвобождения инсулиноподобных пептидов и повышения их уровня в крови. Кодируемые этими генами белки связываются с заостренными концами F-актина и предотвращают полимеризацию и деполимеризацию актиновых филаментов, подавляя активный рост актиновых филаментов и стабилизируя цитоскелет. Гиперэкспрессия этой микроРНК приводит к ингибированию продукции α-синуклеина, действующего как молекулярный шаперон при сборке комплексов белка, ассоциированного с синаптосомами (SNARE-комплексов). В свою очередь, α-синуклеин и SNARE осуществляют слияние внутриклеточных транспортных везикул c мембраной мишенью, ограничивая SNARE-зависимую сборку инсулина и слияние гранул инсулина [11].
MiR-26a экспрессируется в β-клетках островков Лангерганса поджелудочной железы и ингибирует экспрессию зависимой от напряжения субъединицы-C кальциевых каналов L-α1 типа, которая опосредует приток кальция и SNARE-зависимое слияние гранул инсулина с плазматической мембраной β-клеток и последующее высвобождение инсулина в системный кровоток [19].
Продукция монокарбоксилатного транспортера 1 типа (MCT1) в β-клетках островков Лангерганса поджелудочной железы может ингибироваться при гиперэкспрессии трех паралогов miR-29, непосредственно подавляющих матричную РНК гена MCT1. Этот белок-транспортер осуществляет перенос лактата и пирувата в митохондрии. При ингибировании продукции MCT1 β-клетки перестают вырабатывать инсулин во время физических нагрузок, что приводит к значительному повышению уровня циркулирующих лактата и пирувата в системном кровотоке [11].
MiR-130a, miR-130b и miR-152 снижают уровни глюкокиназы (GCK) и субъединицы E1-α1 пируватдегидрогеназы (PDHA1). GCK действует на первом этапе гликолиза. PDHA1 преобразует пируват, полученный в результате гликолиза, в ацетил-КоА в митохондриях. Гиперэкспрессия данных микроРНК снижает внутриклеточное соотношение АТФ/АДФ, синтез и секрецию инсулина [20].
Экспериментальная гиперэкспрессия miR-187 на животной модели приводила к ингибированию протеинкиназы 1 типа, взаимодействующей с гомеодоменом (HIPK1), ослабляя вызванную глюкозой секрецию инсулина in vitro [11]. Экспериментальная гиперэкспрессия miR-200 (на животной модели) приводила к апоптозу β-клеток островков Лангерганса путем подавления экспрессии гена антиапоптоза и стрессоустойчивости DNAJC3 (DnaJ homolog subfamily C member 3), играющего важную роль в развитии сахарного диабета, и гена XIAP (X-chromosome-linked inhibitor of apoptosis protein), ингибитора апоптотической каспазы [21].
MiR-204 подавляет экспрессию рецептора глюкагон-подобного пептида 1 типа (glucagon-like peptide-1, GLP1) на поверхности β-клеток островков Лангерганса поджелудочной железы, тем самым блокируя GLP1-провоцированное высвобождение инсулина из β-клеток путем увеличения их чувствительности к глюкозе [11]. По схожему с miR-7 механизму работает и miR-375, влияющая на экспрессию миотрофина, который связывается со свободным CAPZA1 и регулирует его активность, препятствуя связи CAPZA1 с острым концом F-актина [11].
MiR-802 снижает экспрессию инсулина, блокируя связь промотора гена, кодирующего инсулин, с фактором транскрипции NEUROD1 (neurogenic differentiation 1). Этот фактор активирует транскрипцию генов, содержащих специфическую последовательность ДНК, известную как E-box, и регулирует экспрессию гена инсулина, влияя на риск развития сахарного диабета II типа. Ингибирование секреции инсулина работает по схожему с miR-26a принципу, противодействуя притоку кальция путем подавления рецепторов WNT5A (protein Wnt-5a), действующих аутокринным/паракринным образом и передающих сигналы через Ca²⁺/кальмодулин-зависимую протеинкиназу II типа, стимулирующую активность кальциевого канала высокого напряжения для притока кальция [22].
Регуляция метаболизма глюкозы
Известно, что подавляют гликонеогенез и метаболизм глюкозы miR-7a, miR-26a, miR-27, miR-29, miR-33b, miR-103, miR-107, miR-124, miR-130a, miR-130b, miR-143, miR-152, miR-155, miR-187, miR-200, miR-204, miR-336, miR-375, miR-378, miR-451-1, miR-466b, miR-802.
MiR-27, miR-29 и miR-451-1 ингибируют гликонеогенез в печени путем регуляции функциональной активности глюконеогенного пути (глицеролкиназы, GK) и фактора транскрипции (FOXO1) [11]. MiR-33b непосредственно подавляет экспрессию двух ключевых генов глюконеогенеза, кодирующих фосфоенолпируваткарбоксикиназу 1 (PCK1) и глюкозо-6-фосфатазу (G6PC), что подавляет глюконеогенез [11]. МiR-466b непосредственно ингибирует экспрессию фосфоенолпируваткарбоксикиназы (PEPCK), ключевого глюконеогенетического фермента [11]. Механизмы подавления метаболизма глюкозы у остальных микроРНК освещались ранее [11][16–22].
Регуляция аппетита
Воздействуя на различные структуры головного мозга, а также на множество нижеописанных сигнальных путей, АП первого и новых поколений способны воздействовать на регуляцию аппетита и вызывать расстройство пищевого поведения, связанное с АИМетС [76, 77].
В ходе исследований установлено, что микроРНК участвуют в регуляции аппетита путем нацеливания на нейроны проопиомеланолипокортина (POMC) и нейропептида Y (NPY) в аркуатных ядрах гипоталамуса. В частности, к возбуждающим аппетит микроРНК относятся miR-let7a, miR-7a, miR-9, miR-30e, miR-100, miR-132, miR-141, miR-145, miR-200a, miR-218, miR-342, miR-383, miR-384-3p, miR-429, miR-488.
MiR-7a высоко экспрессируется в нейронах NPY/AgRP, которые оказывают орексигенный эффект [28]. Гиперэкспрессия miR-200a в гипоталамусе связана с нарушением контроля за потреблением пищи и нарушением передачи сигналов лептина/инсулина через изменение экспрессии IRS2. По схожему механизму орексигенный эффект оказывают miR-141 и miR-429 [25]. Голодание приводит к гиперэкспрессии miR-let7a, miR-9, miR-30e, miR-132, miR-145 и miR-218, что свидетельствует об орексигенном эффекте этих микроРНК [27].
При снижении продукции лептина повышается уровень экспрессии miR-383, miR-384-3p и miR-488, что приводит к ингибированию матричной РНК гена POMC, кодирующего ключевой анорексигенный регулятор потребления пищи [25]. MiR-342 связана с увеличением популяции и усиленной активацией NPY-орексигенных нейронов, что приводит к повышению потребления пищи [26] с риском развития центрального ожирения.
Регуляция экспрессии нейропептида Y
Длительный прием некоторых АП (например, клозапина и рисперидона) может приводить к повышению уровня NPY и других биомаркеров АИМетС в сыворотке крови [77][78].
Идентифицированы микроРНК, связанные с увеличением экспрессии NPY. Так, гиперэкспрессия miR-708, miR-2137 в моделях клеток гипоталамуса повышает уровень экспрессии мРНК Npy через косвенный механизм, который не включает прямое связывание с Npy 3’UTR [29].
Регуляция чувствительности к лептину
В последние годы активно обсуждается влияние АП новых поколений на изменение сывороточных уровней протеогормона энергетического обмена лептина и лептиночувствительности адипоцитов. Этот гормон путем воздействия на клеточные рецепторы в дугообразном и вентромедиальном ядрах гипоталамуса участвует в регуляции аппетита, предотвращает гиперфагию и развитие ожирения. Исследования последних лет продемонстрировали, что изменение уровня лептина в крови может рассматриваться как перспективный биомаркер АИМетС у пациентов с РШС [77][79].
Показана отрицательная корреляционная связь между повышением уровня miR-15a, miR-16, miR-33, miR-223, miR-363 и miR-532 и снижением уровня лептина в плазме крови [8]. Кроме того, гиперэкспрессия miR-200a, miR-200b и miR-429 обнаружена в орексигенных нейронах гипоталамуса у нокаутных мышей с первичным дефицитом лептина и первичным дефицитом рецептора лептина (LepR). MiR-200a напрямую ингибирует IRS2 и LepR, которые являются ее прямыми мишенями. Ингибирование miR-200a в гипоталамусе повышает экспрессию мРНК LEPR и IRS2, увеличивая чувствительность орексигенных нейронов гипоталамуса к лептину и инсулину [28].
Регуляция экспрессии орексина
Развитие АИМетС у пациентов с РШС может быть непосредственно связано с влиянием АП на молекулы, участвующие в регуляции системного метаболизма, в частности на нейропептид орексин, синтезирующийся нейронами гипоталамуса [80]. Некоторые микроРНК ингибируют экспрессию орексина, что представляет клинический интерес, поскольку этот нейропептид не только участвует в регуляции цикла сна и бодрствования, вегетативных функций, но и в регуляции пищевого поведения.
В частности, miR-137, miR-637, miR-654 и miR-665 ингибируют мРНК HCRT в нейронах гипоталамуса [19]. При этом исследователи отмечают, что miR-137 генетически связана с повышенным риском не только АИМетС у пациентов с психическими расстройствами, но и непосредственно с риском развития психических расстройств (в частности, шизофрении) [32].
Регуляция экспрессии тиреоидных гормонов
Известно, что длительный прием АП взаимосвязан с изменением экспрессии тиреоидных гормонов и нарушением функции щитовидной железы. При этом большинство ранее проведенных исследований продемонстрировали АП-индуцированное повышение уровня экспрессии тиреотропного гормона [81]. Снижение уровня этих гормонов коррелирует с повышенными концентрациями общего холестерина, ЛПНП и триглицеридов в крови. При этом патогенетическое прогрессирование дислипидемии, связанной с вторичным гипотиреозом, может быть ассоциировано со снижением сывороточной концентрации тиреоидных гормонов и повышением сывороточной концентрации тиреотропного гормона (ТТГ). Таким образом, АП-индуцированный гипотиреоз может вызывать дислипидемию и связанные с ней метаболические расстройства, включая АИМетС [82].
Идентифицированы несколько микроРНК (miR-21, miR-146, miR-214), которые индуцируют экспрессию тиреоидных гормонов. МикроРНК контролируют экспрессию тиреоидных гормонов путем регуляции ферментов семейства дейодиназ. Дейодиназы 1 типа (DIO1) и 2 типа (DIO2) катализируют превращение Т4 в Т3 в тканях-мишенях, повышая внутриклеточный уровень активного гормона. Дейодиназа 3 типа (DIO3) вызывает инактивацию гормонов, поскольку она преобразует T4 (тироксин) и T3 (трийодтиронин) в неактивные метаболиты (rT3 — биологически неактивный изомер тиреоидного гормона Т3; и T2 известный как 3,5-T2 — Janus-faced thyroid hormone metabolite) путем дейодирования по внутреннему кольцу [34]. Кроме того, некоторые микроРНК (miR-27, miR-155, miR-181, miR-200a, miR-221, miR-224, miR-246, miR-383, miR-425) снижают экспрессию тиреоидных гормонов [34]. При этом miR-27, miR-155, miR-181, miR-200a, miR-221, miR-246 и miR-425 снижают экспрессию TRβ, что вместе с регуляцией DIO1 приводит к локальному гипотиреозу и снижению передачи сигналов тиреоидных гормонов. Гиперэкспрессия miR-224 и miR-383 репрессирует мРНК DIO1, что приводит к снижению продукции тиреоидных гормонов [34].
Регуляция экспрессии паратиреоидного гормона
Нарушение метаболизма, ассоциированное с дефицитом витамина D, действует синергически с другими путями, способствуя увеличению веса, индуцированного приемом АП. Хотя прямое катаболическое действие АП на молекулу витамина D не продемонстрировано, АП могут снижать уровень паратиреоидного гормона (ПТГ) в крови за счет нарастания жировой ткани и фолликулярных структур, снижения функциональной активности главных темных паратиреоцитов и усиления функции оксифильных клеток в паращитовидной железе, ухудшать метаболизм витамина D и снижать его уровень [83]. Гиперэкспрессия mir-24 ингибирует продукцию ПТГ, связывая транскрипты CDKN1B/p27, CDKN2A/p16, TGFβ1 и CASP8, которые участвуют в развитии гиперпаратиреоза [35].
В свою очередь, miR-27b ингибирует экспрессию гена VDR (vitamin D receptor), кодирующего ядерный рецептор витамина D, за счет чего снижается чувствительность клеток к витамину D, способствуя развитию вторичного гиперпаратиреоза [35].
Перспективы использования микроРНК как биомаркеров АИМетС
Переход к персонализированной психиатрии отражает современные подходы к индивидуальной оценке безопасности и риска развития нежелательных реакций АП на фоне фармакотерапии психических расстройств [84][85]. Исследование АП-индуцированного изменения уровня экспрессии микроРНК возможно в различных тканях (головной мозг, периферические ткани), но наибольший клинический интерес представляют микроРНК, циркулирующие в периферической крови (плазме, экзосомах, мононуклеарах) в связи с высокой доступностью биообразцов. Например, известно, что в плазме крови рисперидон и арипипразол подавляют экспрессию циркулирующих miR-130b и miR-193a-3p [86], а оланзапин, кветиапин, зипрасидон и рисперидон ингибируют экспрессию miR-30e, miR-132, miR-195 и miR-432 [87]; в мононуклеарах периферической крови — рисперидон, оланзапин, кветиапин, арипипразол и зипрасидон повышают экспрессию miR-30a-5p [88], оланзапин, кветиапин, зипрасидон и рисперидон снижают экспрессию miR-21 [89], а рисперидон повышает экспрессию miR-132, miR-664* и miR-1271 [90].
Проанализированы результаты 34 исследований (9 клинических и 25 доклинических) за 2014–2024 гг., отражающих роль микроРНК в ключевых звеньях патогенеза МетС и АИМетС. Исследования с участием пациентов с МетС в целом и АИМетС в частности у пациентов с психическими расстройствами пока единичны и выполнены на небольших выборках, что не позволяет обобщить представленные результаты и транслировать их в клиническую практику. Однако настоящий обзор демонстрирует потенциальную роль циркулирующих микроРНК как прогностических или предиктивных эпигенетических биомаркеров АИМетС, особенно тех микроРНК, которые участвуют в нескольких звеньях патогенеза АИМетС. Это позволило классифицировать микроРНК в зависимости от степени риска развития АИМетС: низкий риск для микроРНК с сугубо протективными свойствами для двух и более звеньев патогенеза АИМетС; средний риск для микроРНК с предиктивными свойствами в отношении одного звена патогенеза, но протективными свойствами в отношении другого звена; высокий риск для микроРНК-предикторов двух и более звеньев патогенеза АИМетС (рис. 1).
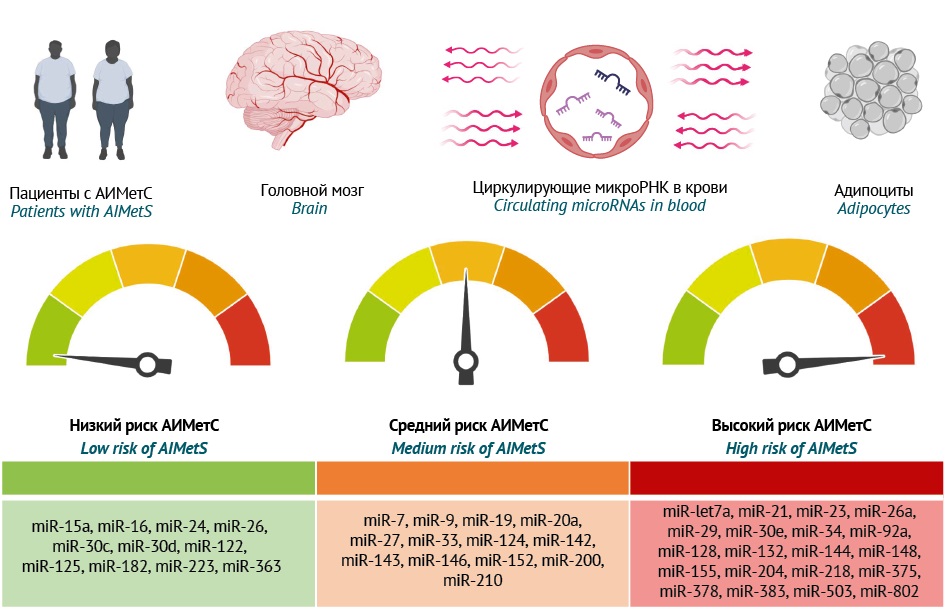
Рисунок подготовлен авторами / The figure is prepared by the authors
Рис. 1. Градация циркулирующих микроРНК в крови по степени риска развития антипсихотик-индуцированного метаболического синдрома (АИМетС)
Fig. 1. Circulating microRNA classification based on the risk of developing antipsychotic-induced metabolic syndrome (AIMetS)
Продолжение изучения этих микроРНК важно для последующего планирования трансляционных исследований, обеспечивающих адаптацию и применение полученных результатов в реальной клинической практике психиатра. Учитывая, что циркулирующие микроРНК являются стабильными молекулами, устойчивыми при пробоподготовке и повторных циклах заморозки-разморозки, исследование их уровня в периферической крови является перспективным в связи с ожидаемой большой повторяемостью полученных результатов у разных пациентов с АИМетС.
В то же время данные некоторых проанализированных нами исследований были противоречивыми. Так, гиперэкспрессия провоспалительной [9] микроРНК (miR-let7а) ассоциировалась с инсулинорезистентностью [11]. Кроме того, эта микроРНК обладала орексигенными свойствами, ассоциированными с повышением чувствительности к лептину [27]. Однако ее паралог (miR-let7b) оказывал ингибирующее действие на экспрессию NPY, что приводило к подавлению аппетита и снижению риска центрального ожирения [29]. МiR-7 индуцирует метаболизм липидов [11] и препятствует развитию системного воспаления [9], но подавляет углеводный метаболизм, снижая экспрессию инсулина [11], и оказывает орексигенный эффект [28]. Противовоспалительная miR-9 [9] также оказывает орексигенный эффект через увеличение чувствительности к лептину [27]. Ингибирующая системное воспаление miR-20а [9] может быть индуктором адипогенеза [10], но два ее паралога (miR-26 и miR-26a) оказывают принципиально разное воздействие на метаболизм глюкозы. МiR-26 активирует экспрессию инсулина и, как следствие, усиливает метаболизм углеводов путем ингибирования репрессоров транскрипции инсулина [23], но ее паралог (miR-26a) ингибирует SNARE-зависимое высвобождение инсулина в системный кровоток [19].
Результаты исследований роли miR-27 и ее паралогов в патогенезе МетС и АИМетС противоречивы. Так, miR-27а ингибирует антиоксидантный путь Nrf2 [6] и способствует развитию системного воспаления [9], а также приводит к накоплению гликогена в скелетных мышцах и миокарде [11]. Однако miR-27a подавляет дифференцировку адипоцитов [10] и регулирует метаболизм липидов в печени, препятствуя развитию НАЖБП совместно с miR-27b [11]. МiR-27 ингибирует адипогенез через активацию Wnt-пути в преадипоцитах 3T3-L1 [10], но при этом снижает выработку глюкозы печенью, воздействуя на ферменты глюконеогенного пути [11], и способствует развитию гипотиреоза [34].
Некоторые исследователи не изучали паралоги, а оценивали роль в патогенезе отдельных звеньев патогенеза МетС и АИМетС семейства в целом, что (по нашему мнению) вносит путаницу и ухудшает понимание полученных результатов. Например, влияние на патогенез МетС оказывают микроРНК семейства miR-33 [8][ 11][ 28]. В целом гипоэкспрессия микроРНК этого семейства приводит к развитию центрального ожирения и инсулинорезистентности. Паралоги miR-33a и miR-33b координированно ингибируют обратный транспорт холестерина с периферии обратно в печень, подавляют ферменты окисления жирных кислот, а также ингибируют выведение холестерина из организма [11]. Гипоэкспрессия miR-33a/b оказывает атеропротекторные эффекты, такие как регуляция функциональной поляризации макрофагов и увеличение транспорта холестерина из макрофагов в плазму, печень и кал, что предотвращает образование пенистых клеток и атеросклероз [8]. Семейство miR-33 снижает чувствительность к инсулину, приводя к развитию инсулинорезистентности. МiR-33 регулирует метаболизм глюкозы, подавляя гликогенолитические ферменты, что приводит к накоплению гликогена в печени, также существуют исследования, демонстрирующие, что семейства miR-33 и miR-33b (в частности) непосредственно подавляют экспрессию ферментов глюконеогенеза [11]. Дополнительно отмечается, что гипоэкспрессия miR-33 (без уточнения паралогов) подавляет аппетит, снижая активность AgRP-нейронов гипоталамуса [24], но при этом снижает чувствительность к лептину [28].
Двойственно на риск развития МетС и АИМетС влияет miR-142. Так, ее противовоспалительный эффект связан с ингибированием синтеза провоспалительных молекул [9], а прооксидантный эффект — со снижением транскрипции антиоксидантного пути Nrf2 [6]. Противовоспалительная miR-143 [9] снижает экспрессию NPY [29], но ассоциирована с усилением адипогенной дифференцировки в преадипоцитах 3T3-L1, центральным ожирением [10] и развитием инсулинорезистентности [11].
Прооксидантная miR-146 [7] оказывает противовоспалительный эффект [9]. Ее паралоги (miR-146а и miR-146b) имеют принципиально разное влияние на адипогенез — miR-146а ингибирует адипогенез, а miR-146b способствует адипогенезу в преадипоцитах 3T3-L1 [10]. МiR-146 повышает уровень тиреоидных гормонов [34], а ее паралог miR-146b повышает экспрессию ПТГ [35].
MiR-152 защищает клетки от окислительного стресса [6], но при этом снижает синтез и секрецию инсулина [20]. Антиоксидантная [6] и противовоспалительная [9] miR-210 является индуктором адипогенеза и биомаркером центрального ожирения [10].
Различия в представленных результатах могут быть обусловлены тем, что проанализированные нами фундаментальные (преимущественно) и клинические исследования имели вариабельный дизайн, а также тем, что в них не учитывались модифицируемые и немодифицируемые факторы риска МетС и АИМетС, как, например, гиподинамия, объем потребления пищи и наследственная предрасположенность.
ЗАКЛЮЧЕНИЕ
Представленный обзор демонстрирует, что чувствительность и специфичность эпигенетических биомаркеров АИМетС могут варьировать в широком диапазоне в зависимости от характера их влияния (предиктивного или протективного) на один или несколько основных звеньев патогенеза рассматриваемой распространенной нежелательной реакции психофармакотерапии, включая окислительный стресс, системное воспаление, регуляцию адипогенеза и развитие центрального ожирения, липидный метаболизм, гомеостаз холестерина ЛПВП и ЛПНП, атерогенез, жировой гепатоз, а также регуляцию чувствительности к инсулину, его экспрессию, метаболизм глюкозы, аппетита, экспрессию нейропептида Y, орексина тиреоидных гормонов, паратиреоидного гормона, чувствительность к лептину. Наиболее изученными являются микроРНК — предиктивные биомаркеры окислительного стресса (miR-1, miR-21, miR-23b, miR-27a, miR-28, miR-29, miR-34a, miR-92a, miR-93, miR-101, miR-106b, miR-128, miR-129, miR-140, miR-142, miR-144, miR-146, miR-148, miR-153, miR-155, miR-181c, miR-193b, miR-320, miR-365, miR-375, miR-383, miR-495, miR-503, miR-802) и системного воспаления (miR-21, miR-23a, miR-27a, miR-29a, miR-34a, miR-34c, miR-92a, miR-132, miR-138, miR-155, miR-200, miR-let7a) у пациентов с высоким риском развития МетС и АИМетС. Перспективными и вызывающими большой научный и клинический интерес предиктивными эпигенетическими биомаркерами АИМетС являются микроРНК, влияющие на уровень экспрессии нейропептидов и чувствительность к ним, включая NPY (miR-let7b, miR-29b, miR-33, miR-140, miR-143, miR-503), лептин (miR-let7a, miR-9, miR-30e, miR-132, miR-145, miR-218, miR-342) и орексин (miR-137, miR-637, miR-654, miR-665).
Направлениями исследований в будущем может быть изучение не только предиктивной роли микроРНК в патогенезе основных звеньев АИМетС, но и комплексного подхода к разработке сигнатур циркулирующих микроРНК, наиболее чувствительных и специфичных у пациентов с высоким риском АИМетС.
Разработка новых панелей эпигенетических биомаркеров этой распространенной нежелательной реакции при применении АП первого и новых поколений будет способствовать повышению безопасности фармакотерапии психических расстройств. Однако для того чтобы транслировать полученные в мире результаты доклинических и клинических исследований в клиническую практику, требуется проведение российских исследований для уточнения данных о чувствительности и специфичности циркулирующих микроРНК в крови как биомаркеров АИМетС.
Вклад авторов. Все авторы подтверждают соответствие своего авторства критериям ICMJE. Наибольший вклад распределен следующим образом: Н.А. Шнайдер — написание текста рукописи и ее доработка по результатам рецензирования; Р.Ф. Насырова — общая концепция, руководство проектом, утверждение окончательной версии рукописи для публикации; Н.А. Пекарец — работа с базами данных, написание текста рукописи; В.В. Гречкина — работа с базами данных, подготовка графических материалов; М.М. Петрова — дизайн исследования, редактирование текста рукописи.
Authors’ contributions. All the authors confirm that they meet the ICMJE criteria for authorship. The most significant contributions were as follows. Natalia А. Shnayder drafted the manuscript and revised it based on the peer-review results. Regina F. Nasyrova developed the general concept of the study, managed the project, and approved the final version of the manuscript for publication. Nikolai A. Pekarets worked with databases and drafted the manuscript. Violetta V. Grechkina worked with databases and prepared illustrations. Marina M. Petrova designed the study and edited the manuscript.
Список литературы
1. Correll CU, Solmi M, Croatto G, et al. Mortality in people with schizophrenia: A systematic review and meta-analysis of relative risk and aggravating or attenuating factors. World Psychiatry. 2022;21(2):248–71. https://doi.org/10.1002/wps.20994
2. Peritogiannis V, Ninou A, Samakouri M. Mortality in schizophrenia-spectrum disorders: Recent advances in understanding and management. Healthcare (Basel). 2022;10(12):2366. https://doi.org/10.3390/healthcare10122366
3. De Hert M, Detraux J, van Winkel R, Yu W, Correll C. Metabolic and cardiovascular adverse effects associated with antipsychotic drugs. Nat Rev Endocrinol. 2012;8(2):114–26. https://doi.org/10.1038/nrendo.2011.156
4. De Hert M, Correll CU, Bobes J, Cetkovich-Bakmas M, Cohen D, Asai I, et al. Physical illness in patients with severe mental disorders. I. Prevalence, impact of medications and disparities in health care. World Psychiatry. 2011;10(1):52–77. https://doi.org/10.1002/j.2051-5545.2011.tb00014.x
5. Khasanova AK, Dobrodeeva VS, Shnayder NA, Petrova MM, Pronina EA, Bochanova EN, et al. Blood and urinary biomarkers of antipsychotic-induced metabolic syndrome. Metabolites. 2022;12(8):726. https://doi.org/10.3390/metabo12080726
6. Saha S. Role of microRNA in oxidative stress. Stresses. 2024;4(2):269–81. https://doi.org/10.3390/stresses4020016
7. Włodarski A, Strycharz J, Wróblewski A, Kasznicki J, Drzewoski J, Śliwińska A. The role of microRNAs in metabolic syndrome-related oxidative stress. Int J Mol Sci. 2020;21(18):6902. https://www.doi.org/10.3390/ijms21186902
8. Carvalho GB, Brandão-Lima PN, Payolla TB, Lucena SEF, Sarti FM, Fisberg RM, Rogero MM. Circulating miRNAs are associated with low-grade systemic inflammation and leptin levels in older adults. Inflammation. 2023;46(6):2132–46. https://www.doi.org/10.1007/s10753-023-01867-6
9. Das K, Rao LVM. The role of microRNAs in inflammation. Int J Mol Sci. 2022;23(24):15479. https://www.doi.org/10.3390/ijms232415479
10. Engin AB, Engin A. Adipogenesis-related microRNAs in obesity. ExRNA. 2022;4:16. https://www.doi.org/10.21037/exrna-22-4
11. Agbu P, Carthew RW. MicroRNA-mediated regulation of glucose and lipid metabolism. Nat Rev Mol Cell Biol. 2021;22(6):425–38. https://www.doi.org/10.1038/s41580-021-00354-w
12. Dong M, Ye Y, Chen Z, Xiao T, Liu W, Hu F. MicroRNA 182 is a novel negative regulator of adipogenesis by targeting CCAAT/enhancer-binding protein α. Obesity (Silver Spring). 2020;28(8):1467–76. https://doi.org/10.1002/oby.22863
13. Wagschal A, Najafi-Shoushtari SH, Wang L, Goedeke L, Sinha S, deLemos AS, et al. Genome-wide identification of microRNAs regulating cholesterol and triglyceride homeostasis. Nat Med. 2015;21(11):1290–7. https://www.doi.org/10.1038/nm.3980
14. Cheng J, Cheng A, Clifford BL, Wu X, Hedin U, Maegdefessel L, et al. MicroRNA-144 silencing protects against atherosclerosis in male, but not female mice. Arterioscler Thromb Vasc Biol. 2020;40(2):412–25. https://www.doi.org/10.1161/ATVBAHA.119.313633
15. Irani S, Iqbal J, Antoni WJ, Ijaz L, Hussain MM. MicroRNA-30c reduces plasma cholesterol in homozygous familial hypercholesterolemic and type 2 diabetic mouse models. J Lipid Res. 2018;59(1):144–54. https://doi.org/10.1194/jlr.M081299
16. Trajkovski M, Hausser J, Soutschek J, Bhat B, Akin A, Zavolan M, et al. MicroRNAs 103 and 107 regulate insulin sensitivity. Nature. 2011;474(7353):649–53. https://doi.org/10.1038/nature10112
17. Liu W, Cao H, Ye C, Chang C, Lu M, Jing Y, et al. Hepatic miR-378 targets p110α and controls glucose and lipid homeostasis by modulating hepatic insulin signalling. Nat Commun. 2014;5:5684. https://doi.org/10.1038/ncomms6684
18. Kornfeld JW, Baitzel C, Könner AC, Nicholls HT, Vogt MC, Herrmanns K, et al. Obesity-induced overexpression of miR-802 impairs glucose metabolism through silencing of Hnf1b. Nature. 2013;494(7435):111–5. https://doi.org/10.1038/nature11793
19. Xu H, Du X, Xu J, Zhang Y, Tian Y, Liu G, et al. Pancreatic β cell microRNA-26a alleviates type 2 diabetes by improving peripheral insulin sensitivity and preserving β cell function. PLoS Biol. 2020;18(2):e3000603. https://doi.org/10.1371/journal.pbio.3000603
20. Ofori JK, Salunkhe VA, Bagge A, Vishnu N, Nagao M, Mulder H, et al. Elevated miR-130a/miR130b/miR-152 expression reduces intracellular ATP levels in the pancreatic beta cell. Sci Rep. 2017;7:44986. https://doi.org/10.1038/srep44986
21. Belgardt BF, Ahmed K, Spranger M, Latreille M, Denzler R, Kondratiuk N, et al. The microRNA-200 family regulates pancreatic beta cell survival in type 2 diabetes. Nat Med. 2015;21(6):619–27. https://doi.org/10.1038/nm.3862
22. Zhang F, Ma D, Zhao W, Wang D, Liu T, Liu Y, et al. Obesity-induced overexpression of miR-802 impairs insulin transcription and secretion. Nat Commun. 2020;11(1):1822. https://doi.org/10.1038/s41467-020-15529-w
23. Melkman-Zehavi T, Oren R, Kredo-Russo S, Shapira T, Mandelbaum AD, Rivkin N, et al. miRNAs control insulin content in pancreatic β-cells via downregulation of transcriptional repressors. EMBO J. 2011;30(5):835–45. https://doi.org/10.1038/emboj.2010.361
24. Price NL, Fernández-Tussy P, Varela L, Cardelo MP, Shanabrough M, Aryal B, et al. microRNA-33 controls hunger signaling in hypothalamic AgRP neurons. Nat Commun. 2024;15(1):2131. https://www.doi.org/10.1038/s41467-024-46427-0
25. Taouis M. MicroRNAs in the hypothalamus. Best Pract Res Clin Endocrinol Metab. 2016;30(5):641–51. https://www.doi.org/10.1016/j.beem.2016.11.006
26. Zhang D, Yamaguchi S, Zhang X, Yang B, Kurooka N, Sugawara R, et al. Upregulation of mir342 in diet-induced obesity mouse and the hypothalamic appetite control. Front Endocrinol (Lausanne). 2021;12:727915. https://www.doi.org/10.3389/fendo.2021.727915
27. Sangiao-Alvarellos S, Pena-Bello L, Manfredi-Lozano M, Tena-Sempere M, Cordido F. Perturbation of hypothalamic microRNA expression patterns in male rats after metabolic distress: Impact of obesity and conditions of negative energy balance. Endocrinology. 2014;155(5):1838–50. https://www.doi.org/10.1210/en.2013-1770
28. Derghal A, Djelloul M, Azzarelli M, Degonon S, Tourniaire F, Landrier JF, et al. MicroRNAs are involved in the hypothalamic leptin sensitivity. Epigenetics. 2018;13(10–11):1127–40. https://doi.org/10.1080/15592294.2018.1543507
29. Mak KWY, He W, Loganathan N, Belsham DD. Bisphenol A Alters the levels of miRNAs that directly and/or indirectly target neuropeptide Y in murine hypothalamic neurons. Genes (Basel). 2023;14(9):1773. https://www.doi.org/10.3390/genes14091773
30. Dobrodeeva VS, Abdyrahmanova AK, Nasyrova RF. Personalized approach to antipsychotic-induced weight gain prognosis. Personalized Psychiatry and Neurology. 2021;1(1):3–10. https://doi.org/10.52667/2712-9179-2021-1-1-3-10
31. Holm A, Possovre ML, Bandarabadi M, Moseholm KF, Justinussen JL, Bozic I, et al. The evolutionarily conserved miRNA-137 targets the neuropeptide hypocretin/orexin and modulates the wake to sleep ratio. Proc Natl Acad Sci USA. 2022;119(17):e2112225119. https://www.doi.org/10.1073/pnas.2112225119
32. Siegert S, Seo J, Kwon EJ, Rudenko A, Cho S, Wang W, et al. The schizophrenia risk gene product miR-137 alters presynaptic plasticity. Nat Neurosci. 2015;18(7):1008–16. https://www.doi.org/10.1038/nn.4023
33. Azhar S, Dong D, Shen WJ, Hu Z, Kraemer FB. The role of miRNAs in regulating adrenal and gonadal steroidogenesis. J Mol Endocrinol. 2020;64(1):R21–R43. https://www.doi.org/10.1530/JME-19-0105
34. Aranda A. MicroRNAs and thyroid hormone action. Mol Cell Endocrinol. 2021;525:111175. https://www.doi.org/10.1016/j.mce.2021.111175
35. Vaira V, Verdelli C, Forno I, Corbetta S. MicroRNAs in parathyroid physiopathology. Mol Cell Endocrinol. 2017;456:9–15. https://www.doi.org/10.1016/j.mce.2016.10.035
36. Шнайдер НА, Насырова РФ, Пекарец НА, Гречкина ВВ, Петрова ММ. Роль паттерна циркулирующих микроРНК в крови в оценке риска развития антипсихотик-индуцированного метаболического синдрома (обзор): часть 1. Безопасность и риск фармакотерапии. 2025. https://doi.org/10.30895/2312-7821-2025-478
37. Dietrich-Muszalska A, Kolodziejczyk-Czepas J, Nowak P. Comparative study of the effects of atypical antipsychotic drugs on plasma and urine biomarkers of oxidative stress in schizophrenic patients. Neuropsychiatr Dis Treat. 2021;17:555–65. https://doi.org/10.2147/NDT.S283395
38. Tsai MC, Liou CW, Lin TK, Lin IM, Huang TL. Changes in oxidative stress markers in patients with schizophrenia: The effect of antipsychotic drugs. Psychiatry Res. 2013;209(3):284–90. https://doi.org/10.1016/j.psychres.2013.01.023
39. Bhandari R, Kaur J, Kaur S, Kuhad A. The Nrf2 pathway in psychiatric disorders: Pathophysiological role and potential targeting. Expert Opin Ther Targets. 2021;25(2):115–39. https://www.doi.org/10.1080/14728222.2021.1887141
40. Fang X, Yu L, Wang D, Chen Y, Wang Y, Wu Z, et al. Association between SIRT1, cytokines, and metabolic syndrome in schizophrenia patients with olanzapine or clozapine monotherapy. Front Psychiatry. 2020;11:602121. https://doi.org/10.3389/fpsyt.2020.602121
41. Ткачев ВО, Меньшикова ЕБ, Зенков НК. Механизм работы сигнальной системы Nrf2/Keap1/ARE (обзор). Биохимия. 2011;76(4):407–22. https://doi.org/10.1134/s0006297911040031
42. Zhang H, Dai S, Yang Y, Wei J, Li X, Luo P, Jiang X. Role of sirtuin 3 in degenerative diseases of the central nervous system. Biomolecules. 2024;13(5):735. https://doi.org/10.3390/biom13050735
43. Yang W, Nagasawa K, Munch C, Xu Y, Satterstrom K, Jeong S, et al. Mitochondrial sirtuin network reveals dynamic Sirt3-dependent deacetylation in response to membrane depolarization. Cell. 2016;167(4):985–1000.e21. https://doi.org/10.1016/j.cell.2016.10.016
44. Lee BJ, Marchionni L, Andrews CE, Norris AL, Nucifora LG, Wu YC, et al. Analysis of differential gene expression mediated by clozapine in human postmortem brains. Schizophr Res. 2017;185:58–66. https://doi.org/10.1016/j.schres.2016.12.017
45. Sommerfeld-Klatta K, Jiers W, Rzepczyk S, Nowicki F, Łukasik-Głębocka M, Świderski P, et al. The effect of neuropsychiatric drugs on the oxidation-reduction balance in therapy. Int J Mol Sci. 2024;25(13):7304. https://doi.org/10.3390/ijms25137304
46. Nasyrova RF, Moskaleva PV, Vaiman EE, Shnayder NA, Blatt NL, Rizvanov AA. Genetic factors of nitric oxide’s system in psychoneurologic disorders. Int J Mol Sci. 2020;21(5):1604. https://doi.org/10.3390/ijms2105160
47. Patel S, Keating BA, Dale RC. Anti-inflammatory properties of commonly used psychiatric drugs. Front Neurosci. 2023;16:1039379. https://doi.org/10.3389/fnins.2022.1039379
48. Al-Amin MM, Nasir Uddin MM, Mahmud Reza H. Effects of antipsychotics on the inflammatory response system of patients with schizophrenia in peripheral blood mononuclear cell cultures. Clin Psychopharmacol Neurosci. 2013;11(3):144–51. https://doi.org/10.9758/cpn.2013.11.3.144
49. Marcinowicz P, Więdłocha M, Zborowska N, Dębowska W, Podwalski P, Misiak B, et al. A meta-analysis of the influence of antipsychotics on cytokines levels in first episode psychosis. J Clin Med. 2021;10(11):2488. https://doi.org/10.3390/jcm10112488
50. Patlola SR, Donohoe G, McKernan DP. Anti-inflammatory effects of 2nd generation antipsychotics in patients with schizophrenia: A systematic review and meta-analysis. J Psychiatr Res. 2023;160:126–36. https://doi.org/10.1016/j.jpsychires.2023.01.042
51. Cohen T, Sundaresh S, Levine F. Antipsychotics activate the TGFβ pathway effector SMAD3. Mol Psychiatry. 2013;18(3):347–57. https://doi.org/10.1038/mp.2011.186
52. Prestwood TR, Asgariroozbehani R, Wu S, Agarwal SM, Logan RW, Ballon JS, et al. Roles of inflammation in intrinsic pathophysiology and antipsychotic drug-induced metabolic disturbances of schizophrenia. Behav Brain Res. 2021;402:113101. https://doi.org/10.1016/j.bbr.2020.113101
53. Melbourne JK, Pang Y, Park MR, Sudhalkar N, Rosen C, Sharma RP. Treatment with the antipsychotic risperidone is associated with increased M1-like JAK-STAT1 signature gene expression in PBMCs from participants with psychosis and THP-1 monocytes and macrophages. Int Immunopharmacol. 2020;79:106093. https://doi.org/10.1016/j.intimp.2019.106093
54. Cheng L, Xia F, Li Z, Shen C, Yang Z, Hou H, et al. Structure, function and drug discovery of GPCR signaling. Mol Biomed. 2023;4(1):46. https://doi.org/10.1186/s43556-023-00156-w
55. Chen CY, Liu HY, Hsueh YP. TLR3 downregulates expression of schizophrenia gene Disc1 via MYD88 to control neuronal morphology. EMBO Rep. 2017;18(1):169–83. https://doi.org/10.15252/embr.201642586
56. Kositsyn YM, de Abreu MS, Kolesnikova TO, Lagunin AA, Poroikov VV, Harutyunyan HS, et al. Towards novel potential molecular targets for antidepressant and antipsychotic pharmacotherapies. Int J Mol Sci. 2023;24(11):9482. https://doi.org/10.3390/ijms24119482
57. Bahmad HF, Daouk R, Azar J, Sapudom J, Teo JCM, Abou-Kheir W, et al. Modeling adipogenesis: Current and future perspective. Cells. 2020;9(10):2326. https://doi.org/10.3390/cells9102326
58. Ghesmati Z, Rashid M, Fayezi S, Gieseler F, Alizadeh E, Darabi M. An update on the secretory functions of brown, white, and beige adipose tissue: Towards therapeutic applications. Rev Endocr Metab Disord. 2024;25(2):279–308. https://doi.org/10.1007/s11154-023-09850-0
59. Li Y, Zhao X, Feng X, Liu X, Deng C, Hu CH. Berberine alleviates olanzapine-induced adipogenesis via the AMPKα-SREBP pathway in 3T3-L1 Cells. Int J Mol Sci. 2016;17(11):1865. https://doi.org/10.3390/ijms17111865
60. Chen CC, Hsu LW, Huang KT, Goto S, Chen CL, Nakano T. Overexpression of Insig-2 inhibits atypical antipsychotic-induced adipogenic differentiation and lipid biosynthesis in adipose-derived stem cells. Sci Rep. 2017;7(1):10901. https://doi.org/10.1038/s41598-017-11323-9
61. Fehsel K, Bouvier ML. Sex-specific effects of long-term antipsychotic drug treatment on adipocyte tissue and the crosstalk to liver and brain in rats. Int J Mol Sci. 2024;25(4):2188. https://doi.org/10.3390/ijms25042188
62. Ma J, Zheng Y, Sun F, Fan Y, Fan Y, Su X, et al. Research progress in the correlation between SREBP/PCSK9 pathway and lipid metabolism disorders induced by antipsychotics. Zhong Nan Da Xue Xue Bao Yi Xue Ban. 2023;48(10):1529–38. https://doi.org/10.11817/j.issn.1672-7347.2023.230029
63. Vantaggiato C, Panzeri E, Citterio A, Orso G, Pozzi M. Antipsychotics promote metabolic disorders disrupting cellular lipid metabolism and trafficking. Trends Endocrinol Metab. 2019;30(3):189–210. https://doi.org/doi:10.1016/j.tem.2019.01.003
64. Delacrétaz A, Vandenberghe F, Glatard A, Dubath C, Levier A, Gholam-Rezaee M, et al. Lipid disturbances in adolescents treated with second-generation antipsychotics: Clinical determinants of plasma lipid worsening and new-onset hypercholesterolemia. J Clin Psychiatry. 2019;80(3):18m12414. https://doi.org/10.4088/JCP.18m12414
65. O’Donnell C, Demler TL, Trigoboff E, Lee C. The impact of high-density lipoprotein cholesterol (HDL-C) levels and risk of movement disorders in patients taking antipsychotics. Innov Clin Neurosci. 2024;21(4–6):27–30. PMCID: PMC11208005
66. Richards-Belle A, Austin-Zimmerman I, Wang B, Zartaloudi E, Cotic M, Gracie C, et al. Associations of antidepressants and antipsychotics with lipid parameters: Do CYP2C19/CYP2D6 genes play a role? A UK population-based study. J Psychopharmacol. 2023;37(4):396–407. https://doi.org/10.1177/02698811231152748
67. Chen CH, Leu SJ, Hsu CP, Pan CC, Shyue SK, Lee TS. Atypical antipsychotic drugs deregulate the cholesterol metabolism of macrophage-foam cells by activating NOX-ROS-PPARγ-CD36 signaling pathway. Metabolism. 2021;123:154847. https://doi.org/10.1016/j.metabol.2021.154847
68. Gangopadhyay A, Ibrahim R, Theberge K, May M, Houseknecht KL. Non-alcoholic fatty liver disease (NAFLD) and mental illness: Mechanisms linking mood, metabolism and medicines. Front Neurosci. 2022;16:1042442. https://doi.org/10.3389/fnins.2022.1042442
69. Chen J, Huang XF, Shao R, Chen C, Deng C. Molecular mechanisms of antipsychotic drug-induced diabetes. Front Neurosci. 2017;11:643. https://doi.org/10.3389/fnins.2017.00643
70. Weeks KR, Dwyer DS, Aamodt EJ. Antipsychotic drugs activate the C. elegans Akt pathway via the DAF-2 insulin/IGF-1 receptor. ACS Chem Neurosci. 2010;1(6):463–73. https://doi.org/10.1021/cn100010p
71. Kowalchuk C, Kanagasundaram P, Belsham DD, Hahn MK. Antipsychotics differentially regulate insulin, energy sensing, and inflammation pathways in hypothalamic rat neurons. Psychoneuroendocrinology. 2019;104:42–8. https://doi.org/10.1016/j.psyneuen.2019.01.029
72. Li YZ, Di Cristofano A, Woo M. Metabolic role of PTEN in insulin signaling and resistance. Cold Spring Harb Perspect Med. 2020;10(8):a036137. https://doi.org/10.1101/cshperspect.a036137
73. Ramasubbu K, Devi Rajeswari V. Impairment of insulin signaling pathway PI3K/Akt/mTOR and insulin resistance induced AGEs on diabetes mellitus and neurodegenerative diseases: A perspective review. Mol Cell Biochem. 2023;478(6):1307–24. https://doi.org/10.1007/s11010-022-04587-x
74. Zhou W, Sun J, Huai C, Liu Y, Chen L, Yi Z, et al. Multi-omics analysis identifies rare variation in leptin/PPAR gene sets and hypermethylation of ABCG1 contribute to antipsychotics-induced metabolic syndromes. Mol Psychiatry. 2022;27(12):5195–205. https://doi.org/10.1038/s41380-022-01759-5
75. Nwosu BU, Meltzer B, Maranda L, Ciccarelli C, Reynolds D, Curtis L, et al. A potential role for adjunctive vitamin D therapy in the management of weight gain and metabolic side effects of second-generation antipsychotics. J Pediatr Endocrinol Metab. 2011;24(9–10):619–26. https://doi.org/10.1515/jpem.2011.300
76. Mukherjee S, Skrede S, Milbank E, Andriantsitohaina R, López M, Fernø J. Understanding the effects of antipsychotics on appetite control. Front Nutr. 2022;8:815456. https://doi.org/10.3389/fnut.2021.815456
77. Добродеева ВС, Шнайдер НА, Миронов КО, Насырова РФ. Фармакогенетические маркеры антипсихотик-индуцированного набора веса: система лептина и нейропептида Y. Обозрение психиатрии и медицинской психологии имени В.М. Бехтерева. 2021;55(1):3–10. https://doi.org/10.31363/2313-7053-2021-1-3-10
78. Klemettilä JP, Kampman O, Solismaa A, Lyytikäinen LP, Seppälä N, Viikki M, et al. Association study of arcuate nucleus neuropeptide Y neuron receptor gene variation and serum Npy levels in clozapine treated patients with schizophrenia. European Psychiatry. 2017;40:13–9. https://doi.org/10.1016/j.eurpsy.2016.07.004
79. Zhang Y, Li X, Yao X, Yang Y, Ning X, Zhao T, et al. Do leptin play a role in metabolism-related psychopathological symptoms? Front Psychiatry. 2021;12:710498. https://doi.org/10.3389/fpsyt.2021.710498
80. Chen PY, Chang CK, Chen CH, Fang SC, Mondelli V, Chiu CC, et al. Orexin-a elevation in antipsychotic-treated compared to drug-free patients with schizophrenia: A medication effect independent of metabolic syndrome. J Formos Med Assoc. 2022;121(11):2172–81. https://doi.org/10.1016/j.jfma.2022.03.008
81. Burghardt KJ, Mando W, Seyoum B, Yi Z, Burghardt PR. The effect of antipsychotic treatment on hormonal, inflammatory, and metabolic biomarkers in healthy volunteers: A systematic review and meta-analysis. Pharmacotherapy. 2022;42(6):504–13. https://doi.org/10.1002/phar.2689
82. Su X, Chen X, Peng H, Song J, Wang B, Wu X. Novel insights into the pathological development of dyslipidemia in patients with hypothyroidism. Bosn J Basic Med Sci. 2022;22(3):326–39. https://doi.org/10.17305/bjbms.2021.6606
83. Nwosu BU, Meltzer B, Maranda L, Ciccarelli C, Reynolds D, Curtis L, et al. A potential role for adjunctive vitamin D therapy in the management of weight gain and metabolic side effects of second-generation antipsychotics. J Pediatr Endocrinol Metab. 2011;24(9–10):619–26. https://doi.org/10.1515/jpem.2011.300
84. Neznanov NG. A paradigm shift to treat psychoneurological disorders. Personalized Psychiatry and Neurology. 2021;1(1):1–2.
85. Ashurov ZS. The evolution of personalized psychiatry. Personalized Psychiatry and Neurology. 2023;3(2):1–2.
86. Wei H, Yuan Y, Liu S, Wang C, Yang F, Lu Z, et al. Detection of circulating miRNA levels in schizophrenia. Am J Psychiatry. 2015;172(11):1141–7. https://doi.org/10.1176/appi.ajp.2015.14030273
87. Liu S, Zhang F, Shugart YY, Yang L, Li X, Liu Z, et al. MiR143-3p-mediated NRG-1-dependent mitochondrial dysfunction contributes to olanzapine resistance in refractory schizophrenia. Biol Psychiatry. 2022;92(5):419–33. https://doi.org/10.1016/j.biopsych.2022.03.012
88. Liu S, Zhang F, Shugart YY, Yang L, Li X, Liu Z, et al. The early growth response protein 1-miR-30a-5p-neurogenic differentiation factor 1 axis as a novel biomarker for schizophrenia diagnosis and treatment monitoring. Transl Psychiatry. 2017;7(1):e998. https://doi.org/10.1038/tp.2016.268
89. Chen SD, Sun XY, Niu W, Kong LM, He MJ, Fan HM, et al. A preliminary analysis of microRNA-21 expression alteration after antipsychotic treatment in patients with schizophrenia. Psychiatry Res. 2016;244:324–32. https://doi.org/10.1016/j.psychres.2016.04.087
90. Yu HC, Wu J, Zhang HX, Zhang GL, Sui J, Tong WW, et al. Alterations of miR-132 are novel diagnostic biomarkers in peripheral blood of schizophrenia patients. Prog Neuropsychopharmacol Biol Psychiatry. 2015;63:23–9. https://doi.org/10.1016/j.pnpbp.2015.05.007
Об авторах
Н. А. ШнайдерРоссия
Шнайдер Наталья Алексеевна, д-р мед. наук, профессор
ул. Бехтерева, д. 3, Санкт-Петербург, 192019;
ул. Партизана Железняка, д. 1, Красноярск, 660022
Р. Ф. Насырова
Россия
Насырова Регина Фаритовна, д-р мед. наук
ул. Бехтерева, д. 3, Санкт-Петербург, 192019;
пр. Ленина, д. 92, г. Тула, 300012
Н. А. Пекарец
Россия
Пекарец Николай Александрович
ул. Бехтерева, д. 3, Санкт-Петербург, 192019
В. В. Гречкина
Россия
Гречкина Виолетта Владимировна
ул. Бехтерева, д. 3, Санкт-Петербург, 192019
М. М. Петрова
Россия
Петрова Марина Михайловна, д-р мед. наук, профессор
ул. Партизана Железняка, д. 1, Красноярск, 660022
Дополнительные файлы
Рецензия
Для цитирования:
Шнайдер Н.А., Насырова Р.Ф., Пекарец Н.А., Гречкина В.В., Петрова М.М. Циркулирующие микроРНК — перспективные биомаркеры для оценки риска развития антипсихотик-индуцированного метаболического синдрома (обзор): часть 2. Безопасность и риск фармакотерапии. 2025;13(4):394-410. https://doi.org/10.30895/2312-7821-2025-499
For citation:
Shnayder N.A., Nasyrova R.F., Pekarets N.A., Grechkina V.V., Petrova M.M. Circulating MicroRNAs Are Promising Biomarkers for Assessing the Risk of Antipsychotic-Induced Metabolic Syndrome (Review): Part 2. Safety and Risk of Pharmacotherapy. 2025;13(4):394-410. https://doi.org/10.30895/2312-7821-2025-499
JATS XML